Revue TELECOM 193 - Les technologies numériques démocratisent l'analyse génomique
LES TECHNOLOGIES NUMERIQUES DÉMOCRATISENT L'ANALYSE GENOMIQUE
Par Jean-Marc Holder dans la revue TELECOM n° 193
La médecine a toujours été à l’écoute de l’innovation et prompte à adopter de nouvelles technologies en vue d’améliorer la prise en charge des patients. Aujourd’hui, une de ces innovations est en train de révolutionner l’essence même de l’approche médicale : la médecine personnalisée qui vise à adapter les traitements aux caractéristiques propres à chaque patient. Une approche résumée par le leitmotiv : « le bon traitement au bon moment pour chaque patient » (voir encart dédié). La génomique présente le potentiel de devenir un des outils clés de cette nouvelle approche pour deux raisons : sa capacité à révéler les facteurs qui rendent chaque patient unique au niveau le plus fondamental et son rôle direct dans de nombreuses maladies rares et cancers.
Aujourd'hui, l'utilisation de la génomique est facilitée grâce à des évolutions technologiques connues sous le nom de Next Generation Sequencing (NGS) qui ont drastiquement réduit les coûts du séquençage, de deux milliards d’euros il y a 20 ans à quelques centaines d’euros aujourd’hui, tout en simplifiant la lecture de l’information génétique d’un patient. Grâce à cette évolution, les médecins peuvent disposer d’un niveau d’informations sur leurs patients jusqu’ici inespéré. Ces giga-octets de données contiennent ainsi et dans beaucoup de cas les informations permettant de mieux soigner voire de prévenir une maladie chez un patient donné.
Pourtant et malgré ce potentiel, le recours à la génomique reste relativement limité. Un frein à l’adoption qui peut être résolu grâce aux technologies numériques, et en particulier celles de l’intelligence artificielle (IA) et du big data.
Les défis d’un immense volume de données à analyser
Le séquençage du génome d’un seul patient peut engendrer plus de 500 Go d’informations soit l'équivalent d’un disque dur d’un ordinateur personnel. Une fois séquencé, il est nécessaire d’analyser ce vaste amas de données en le comparant à un génome de référence afin d’identifier et séparer les mutations potentiellement pathogènes des variances génétiques normales entre individus.
Parmi ces millions de mutations présentes chez tout individu, l’enjeu est alors d’identifier les quelques mutations qui sont responsables de la maladie dont souffre le patient. C’est à partir de là qu’on peut déduire les informations qui pourront être utiles au médecin pour établir son diagnoctic et décider d’une stratégie de traitement.
En outre, tout ce processus doit être réalisé dans un parfait respect de la confidentialité des données du patient et dans un temps suffisamment court pour que le patient puisse bénéficier des résultats au plus vite.
Face à ce volume, les approches historiques ont consisté à écarter les mutations sans lien évident avec la maladie du patient pour aboutir à un volume de données “traitable”. Cependant cette approche présente un sérieux handicap. En effet, certaines maladies sont influencées par plusieurs gènes aux effets indirects et donc non directement liés à une maladie. En les écartant, il devient impossible de constater la présence systématique d’une mutation jugée sans pertinence alors qu’elle pourrait mener à de nouvelles découvertes et au traitement adéquat.
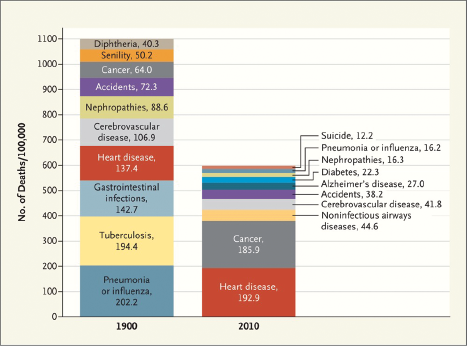
La médecine personnalisée Depuis l’ère d'Hippocrate, la médecine a défini des consignes de traitement par pathologie. Cette approche a démontré son efficacité avec des innovations tel que les antibiotiques qui ont drastiquement réduit le nombre de décès dû à des maladies transmissibles. Aujourd'hui, la majorité des décès est due à des maladies complexes telles que les maladies cardio-vasculaires et les cancers.
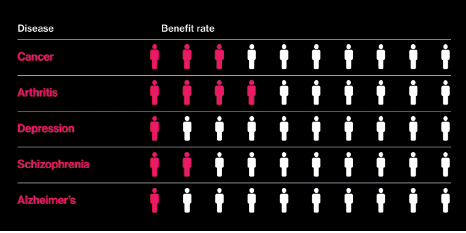
Cependant, dans le paysage des maladies complexes, on constate une certaine perte d’efficacité des approches thérapeutiques. L’illustration ci-dessous démontre la proportion de patients qui présentent une amélioration significative après avoir été traités par le médicament le plus vendu dans chaque catégorie.
L’explication de cette efficacité réduite est simple : les maladies contagieuses, provoquées par un facteur unique qui se transmet d’un individu à un autre, se prêtent à un traitement universel. Les maladies complexes sont quant à elles hétérogènes et peuvent, en dépit de symptômes similaires, être provoquées par des facteurs différents. Il est donc nécessaire de traiter les patients en tant qu’individu pour comprendre les caractéristiques qui lui sont propres et développer un traitement le mieux adapté. C’est le concept de la médecine de précision. Armé de cette information, le corps médical peut chercher des similitudes infimes entre les patients qui répondent à un traitement spécifique et ainsi le prescrire à ceux qui présentent les mêmes caractéristiques à un moment donné. |
L’analyse génomique est rendue plus accessible grâce au big data et à l’IA
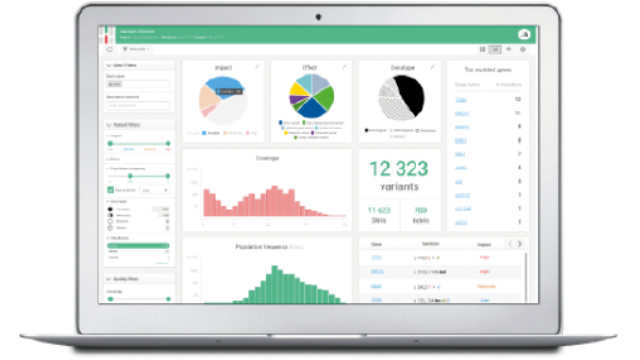
Pour répondre à ces défis, SeqOne a développé un outil d’aide à la décision au biologiste et au médecin en simplifiant l’analyse génomique tout en la rendant plus accessible et plus rapide. Cette solution est accessible sur un Cloud via un simple navigateur internet.
L’enjeu technique de conserver l’intégralité de l’information génomique (Whole Genome Sequencing) a été rendu possible grâce aux technologies du Big Data (Cassandra, Spark, Elastic Search).
C’est l’IA qui permet ensuite de rendre analysable le volume de mutations et d’identifier celles pertinentes pour la maladie d’un patient. Pour cela, la solution de SeqOne intègre non seulement les mutations détectées chez le patient mais aussi ses métadonnées (âge, sexe, symptomes,...) ainsi que les informations connues et partagées au sein de la communauté médicale sur chaque mutation identifiée. Ce modèle d’IA de type “random forrest” apprend grâce aux analyses précédentes de l’utilisateur permettant ainsi une une amélioration continue de la performance et de l’analyse proposée.
Bien que l’IA soit essentielle pour une exploitation efficace de la génomique dans la médecine, il est important de bien choisir la façon de la mettre en œuvre entre un modèle “déterministe” offrant une visibilité sur les inférences qui ont mené au résultat et un modèle “non déterministe” s’apparentant à une “boîte noire” sans justification de la réponse apportée. Pour des raisons de déontologie, nous restons convaincus que dans le domaine de la médecine, l’IA doit rester fermement dans le camp déterministe afin que l'équipe soignante puisse comprendre le raisonnement qui a mené au résultat afin de pouvoir juger de sa pertinence.
Vers une réelle médecine de précision : les perspectives futures
Le prochain enjeu sera de prendre en considération des analyses et des données d’un grand nombre de personnes en vue d’améliorer les modèles d’apprentissage et in fine les diagnostics et les réponses de chaque patient aux traitements reçus pour chaque maladie. Cette mise en commun grandissante permettra ainsi d’établir avec une grande précision et selon le profil d’un patient la thérapie qui lui sera la mieux adaptée.
Ce défi de la montée en charge permettant d’augmenter le nombre de patients analysés peut être résolu grâce à une autre technique de big data, le “edge computing”. À la différence d’un système « pur SaaS» intégralement hébergé dans le Cloud, SeqOne offre la possibilité d'adjoindre un module de calcul décentralisé situé au plus près de la production de données avec un séquenceur qui convertit les échantillons biologiques en données numériques. Cette option permet à la fois d’automatiser l’import des donnés brut, de réduire les temps de transfert des donnés et de minimiser les données sensibles qui transitent sur le réseau. Le résultat : une mise à disposition d’un bilan plus rapide, une meilleure optimisation de l'utilisation du réseau et une sécurité accrue.
Si cette ouverture des données permet d’améliorer non seulement la prise en charge des maladies les plus graves mais encore la prévention et l’identification des patients à risque, une démarche de mise en commun soulève d’énormes enjeux de confidentialité et de données. En effet, la donnée génomique contient un tel nombre d’informations sur un patient que son identification ouvre la voie à de nombreuses dérives. Comment s’assurer que les données génomiques d’un patient ne seront pas utilisées pour le discriminer sur la base de condition médicale ? Des évolutions qui nous obligent aussi à repenser un grand nombre d’éléments centraux en médecine. Quels protocoles de traitement des patients ? Quel rôle et pouvoir décisionnel accorder au patient dans la gestion de la santé ? Quelles politiques déployées concernant l’exploitation des données ?...
C’est en trouvant le juste compromis et les bonnes réponses à ces questions qu’une nouvelle ère de médecine, personnalisée et de précision, s’ouvre à nous.
Biographie de l'auteur
 Jean-Marc Holder, citoyen britannique et trinidadien, travaille depuis longtemps dans le domaine de l'innovation. Après des études en ingénierie et en administration des affaires à l'Université McGill au Canada et à l'Université du Michigan aux États-Unis, il se lance dans les technologies numériques en travaillant avec des entreprises novatrices comme Apple avant d’attraper le virus « start-up ». Il en co-fonde plusieurs dans des domaines aussi divers que les voyages en ligne, la cartographie numérique et les médias sociaux avant de rejoindre l'équipe qui a créé SeqOne Genomics. Jean-Marc vit à Sète dans le sud de la France et est un marin passionné ayant participé aux Jeux Olympiques d'été de 1984 représentant Trinidad et Tobago.
Jean-Marc Holder, citoyen britannique et trinidadien, travaille depuis longtemps dans le domaine de l'innovation. Après des études en ingénierie et en administration des affaires à l'Université McGill au Canada et à l'Université du Michigan aux États-Unis, il se lance dans les technologies numériques en travaillant avec des entreprises novatrices comme Apple avant d’attraper le virus « start-up ». Il en co-fonde plusieurs dans des domaines aussi divers que les voyages en ligne, la cartographie numérique et les médias sociaux avant de rejoindre l'équipe qui a créé SeqOne Genomics. Jean-Marc vit à Sète dans le sud de la France et est un marin passionné ayant participé aux Jeux Olympiques d'été de 1984 représentant Trinidad et Tobago.
"Retour au sommaire de la revue 193"